酶

葡糖苷酶能催化一分子麥芽糖轉化爲兩分子葡萄糖的反應。圖中活性位點以紅色表示,麥芽糖以黑色表示,輔酶NAD以黃色表示(PDB 1OBB)
生物化学的一部分 |
| 生物化学 |
|---|
 |
关键部分 |
|
|
历史和主题 |
|
|
|
|
主题: 生物学, 分子与细胞生物学 |
酶(英语:Enzyme(/ˈɛnzaɪm/ ))是一类大分子生物催化劑。酶能加快化學反應的速度(即具有催化作用)。由酶催化的反應中,反應物稱爲底物,生成的物質稱爲產物。幾乎所有細胞內的代謝過程都離不開酶。酶能大大加快這些過程中各化學反應進行的速率,使代謝產生的物質和能量能滿足生物體的需求[1]:8.1。細胞中酶的類型對可在該細胞中發生的代謝途徑的類型起決定作用。對酶進行研究的學科稱爲「酶學」(enzymology)。
目前已知酶可以催化超過5000種生化反應[2]。大部分酶是蛋白質,有少部分酶是具有催化活性的RNA分子,这些酶被称为核酶。酶的特異性是由其獨特的三級結構決定的。
和所有的催化劑一樣,酶通過降低反應活化能加快化學反應的速率。一些酶可以將底物轉化爲產物的速率提高數百萬倍。一個比較極端的例子是乳清苷-5'-磷酸脫羧酶。該酶可以使在無催化劑條件下需要進行數百萬年的化學反應在幾毫秒內完成[3][4]。從化學原理上講,酶和其它所有催化劑一樣,反應不會使其物質量發生變化。酶亦不能改變化學平衡,這一點和其它催化劑也是一樣的。酶和其它催化劑的不同之處在於,它們的專一性要強得多。一些分子可以影響酶的活性。如酶抑制劑能降低酶的活性,酶激活劑能提高酶的活性。許多藥物及毒物是酶的抑制劑。當超出適宜的溫度和pH值後,酶的活性會顯著下降。
酶在工业和人们的日常生活中的应用也非常广泛。例如,药厂用特定的合成酶来合成抗生素;洗衣粉中添加酶能加速附着在衣物上的蛋白质、淀粉或脂肪漬的分解;嫩肉粉中加入木瓜蛋白酶能將蛋白質分解爲稍小的分子,使肉的口感更嫩滑。
目录
1 发现及研究史
2 命名規則
3 結構
4 機制
4.1 底物結合
4.1.1 「鑰匙和鎖」模型
4.1.2 誘導契合模型
4.2 催化机理
4.3 动态结构
4.4 别构调节
5 輔助因子
5.1 輔酶
6 热力学
7 动力学
8 抑制作用
8.1 可逆抑制作用
8.1.1 竞争性抑制作用
8.1.2 非竞争性抑制作用
8.1.3 反竞争性抑制作用
8.1.4 复合抑制作用
8.2 不可逆抑制作用
8.3 抑制剂的用途
9 生物学功能
9.1 代谢
9.2 活性控制
9.3 相关疾病
10 工业應用
11 參見
12 參考
13 拓展閱讀
发现及研究史

法国科学家路易·巴斯德
酶的发现来源于人们对发酵机理的逐渐了解。早在18世纪末和19世纪初,人们就认识到食物在胃中被消化,[5]用植物的提取液可以将淀粉转化为糖,但对于其对应的机理则并不了解。[6]
到了19世纪中叶,法国科学家路易·巴斯德对蔗糖转化为酒精的发酵过程进行了研究,认为在酵母细胞中存在一种活力物质,命名为“酵素”(ferment)。他提出发酵是这种活力物质催化的结果,并认为活力物质只存在于生命体中,细胞破裂就会失去发酵作用。[7]
1878年,德国生理学家威廉·屈内首次提出了酶(enzyme)这一概念。随后,酶被用于专指胃蛋白酶等一类非活体物质,而酵素(ferment)则被用于指由活体细胞产生的催化活性。

德国科学家爱德华·比希纳
这种对酶的错误认识很快得到纠正。1897年,德国科学家爱德华·比希纳开始对不含细胞的酵母提取液进行发酵研究,通过在柏林洪堡大学所做的一系列实验最终证明发酵过程并不需要完整的活细胞存在。[8]他将其中能够发挥发酵作用的酶命名为发酵酶(zymase)。[9]这一贡献打开了通向现代酶学与现代生物化学的大门,其本人也因“发现无细胞发酵及相应的生化研究”而获得了1907年的诺贝尔化学奖。在此之后,酶和酵素两个概念合二为一,并依据比希纳的命名方法,酶的发现者们根据其所催化的反应将它们命名。通常酶的英文名称是在催化底物或者反应类型的名字最后加上-ase的后缀,而对应中文命名也采用类似方法,即在名字最后加上“酶”。例如,乳糖酶(lactase)是能够剪切乳糖(lactose)的酶;DNA聚合酶(DNA polymerase)能够催化DNA聚合反应(polymerization)。
人们在认识到酶是一类不依赖于活体细胞的物质后,下一步工作就是鉴定其生化组成成分。许多早期研究者指出,一些蛋白质与酶的催化活性相关;但包括诺贝尔奖得主里夏德·维尔施泰特在内的部分科学家认为酶不是蛋白质,他们辩称那些蛋白质只是酶分子的携带者,蛋白质本身并不具有催化活性。1926年,美国生物化学家詹姆斯·萨姆纳完成了一个决定性的实验。他首次从刀豆得到尿素酶结晶,并证明了尿素酶的蛋白质本质。其后,萨姆纳在1931年在过氧化氢酶的研究中再次证实了酶为蛋白质。约翰·霍华德·诺思罗普和温德尔·梅雷迪思·斯坦利通过对胃蛋白酶、胰蛋白酶和胰凝乳蛋白酶等消化性蛋白酶的研究,最终确认蛋白质可以是酶。以后陆续发现的两千余种酶均证明酶的化学本质是蛋白质。以上三位科学家因此获得1946年度诺贝尔化学奖。[10]
由于蛋白质可以结晶,通过X射线晶体学就可以对酶的三维结构进行研究。第一个获得结构解析的酶分子是溶菌酶,一种在眼泪、唾液和蛋清中含量丰富的酶,其功能是溶解细菌外壳。溶菌酶结构由大卫·菲利浦(David Phillips)所领导的研究组解析,并于1965年发表。[11]这一成果的发表标志着结构生物学研究的开始,高分辨率的酶三维结构使得对于酶在分子水平上的工作机制的了解成为可能。
1980年代,托马斯·切赫和悉尼·奥尔特曼分别从四膜虫的rRNA前体的加工研究和细菌的核糖核酸酶P复合物的研究中都发现RNA本身具有自我催化作用,并提出了核酶的概念。这是第一次发现蛋白质以外的具有催化活性的生物分子。
1989年,其二人也因此获得诺贝尔化学奖。[12]
命名規則
酶的命名是衍生自其受質或是要催化的化學反應,在字尾會加上-ase[1]:8.1.3。例如乳糖酶(lactase)、醇脫氫酶(alcohol dehydrogenase)及DNA聚合酶(DNA polymerase)。但有些化學反應可以由幾種不同的酶催化,這些酶稱為同工酶[1]:10.3,而上述的命名法無法處理同工酶的情形。
國際生物化學與分子生物學聯盟提出了酶的命名法,也就是EC編號。每一個酶用一個四位數的數字表示,前面再加上"EC"。第一位數字是酶依酶促反應的機制來分類[13]。
依照第一位數字,可以分為以下六類:
- EC 1 氧化還原酶:催化氧化還原反應的酶類,例如乳酸去氫酶、琥珀酸去氫酶、細胞色素氧化酶、過氧化氫酶、過氧化物酶等。
- EC 2 轉移酶:轉移官能团(例如甲基或是磷酸基團)的酶類,例如甲基轉移酶、氨基轉移酶、己糖激酶、磷酸化酶等。
- EC 3 水解酶:催化受質發生水解反應的酶類,例如澱粉酶、蛋白酶、脂肪酶、磷酸酶等。
- EC 4 裂合酶:用氧化及水解反應以外的方式移去基團的酶類,例如碳酸酐酶、醛縮酶、檸檬酸合酶等。
- EC 5 異構酶:催化分子同分異構反應的酶類,例如磷酸丙糖異構酶、消旋酶等。
- EC 6 連接酶:用共价键結合二個分子的酶類,例如谷氨醯胺合成酶、丙酮酸羧化酶等。。
上述分類會再依底物、生成物及化學結構來分類。用四位數字可以完整的描述一個酶。例如,己糖激酶(EC 2.7.1.1)是轉移酶(EC 2),會將磷酸基團(EC 7)加到六碳糖中,是一個含有醇基的分子(EC 2.7.1)[14]。
結構
@media all and (max-width:720px).mw-parser-output .tmulti>.thumbinnerwidth:100%!important;max-width:none!important.mw-parser-output .tmulti .tsinglefloat:none!important;max-width:none!important;width:100%!important;text-align:center
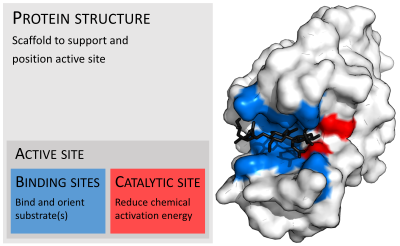
酶結構的組織(以溶菌酶爲例)。圖中結合位點以藍色表示,催化位點以紅色表示,底物肽聚糖以黑色表示(PDB 9LYZ)

酶大都是球蛋白,以單體或聚成複合物對反應進行催化。和其他的蛋白質一樣,酶的三維結構是通過多肽鏈摺疊形成的。氨基酸的序列(一級結構)能決定蛋白質的三維結構,進而影響酶的催化活性[15]。儘管結構決定功能是一條具普適性的規則,一種新的酶的活性不能僅僅通過其結構預測[16]。加熱時或與化學變性劑接觸時,酶結構會發生去摺疊(即變性),原有的結構被打亂,活性也往往隨之喪失[17]。在溫度超過正常水平時,酶就會變性。因此,不難推斷生活在火山環境(比如熱泉)中的細菌的酶具有很強的耐熱性。這些酶使高溫條件下酶促反應的發生成爲可能,在工業上具有很高的利用價值。
酶通常比底物大得多。酶的肽鏈長度從62個氨基酸殘基的4-草酰巴豆酸異構酶的單體[18]到長度超過2,500個氨基酸殘基的動物脂肪酸合酶[19]。酶的結構只有一小部分(大約2-4個氨基酸)是直接與催化相關的。這部分稱爲催化位點(catalytic site)[20]。催化位點通常與一個或多個與底物結合的結合位點(binding site)相連。催化位點與結合位點共同組成了酶的活性位點(active site)。酶的其餘部分起維持活性位點準確的方向以及動力學特性的作用[21]。
在一些酶中,催化與任何一個氨基酸都沒有關係。這類酶另有與催化輔助因子結合的位點[21]。一些酶亦可能包含變構位點。小分子與變構位點的結合可使酶發生構象改變,進而使酶的活性降低或升高[22]。
一些具有生物催化活性的RNA分子稱爲核酶(ribozyme)。這類分子可能單獨發揮催化作用,也可能在與蛋白質結合成複合物的條件下發揮催化作用。最常見的核酶應是核糖體。核糖體是蛋白質以及具有催化活性的RNA的複合物。核糖體的活性位點完全由RNA組成,而蛋白質僅起支架的作用[1]:2.2[23]:695-701。
機制
底物結合
酶在催化化學反應前必須要與底物結合。酶具有很強的專一性,通常僅能與寥寥數種底物結合,催化一種或幾種反應。專一性通過結合區的形狀、電荷、疏水/親水性與底物互補實現。因此,酶可以用來區分化學選擇性上、區域選擇性上、立體專一性上有所不同的結構相似的分子[24]。
一些與基因組複製與表達相關的酶具有很高的專一性和準確性,表现在一些酶所具有的校對機制。以DNA聚合酶爲例,這類酶先催化DNA鏈的合成,再檢查新加上的鹼基是否正確[25]。校對機制確保了酶的極高準確性,哺乳動物的高保真DNA聚合酶在每一億次反應中才會出一次錯誤[1]:5.3.1。RNA聚合酶、氨酰-tRNA合成酶[26]、核糖體[27]也有與DNA聚合酶類似的校對機制[28]。
相对应地,另外一些酶则表現出多底物特异性(enzyme promiscuity)的現象。這類酶專一性弱,能與一系列生理上相關的底物反應。很多此类酶偶然还會出現一定程度的副反應活性,而这可能是進化出新功能酶的起點[29][30]。
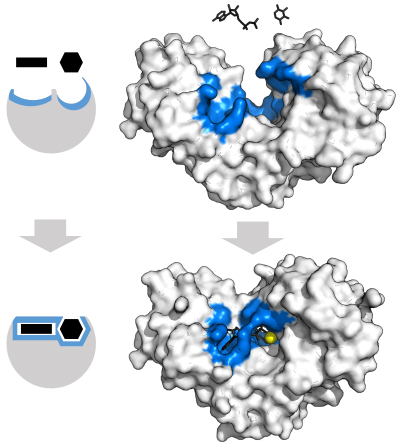
在與底物發生誘導契合後,酶改變形狀,生成酶—底物複合物。己糖激酶的誘導契合運動較大,能包覆住底物ATP和木糖。圖中結合位點用藍色表示,底物用黑色表示,Mg2+輔助因子用黃色表示。(PDB 2E2N, 2E2Q)
「鑰匙和鎖」模型
爲了解釋觀察到的酶的特異性,1894年,赫尔曼·埃米尔·费歇尔提出,酶和底物靠着互補的幾何形狀精準地結合在一起[31]。這一理論即通常所說的「鑰匙和鎖」模型[1]:8.3.2。這一早期的理論解釋了酶的專一性,但卻沒能解釋酶的過渡態爲何能穩定存在[32]。
誘導契合模型
1958年,丹尼爾·科甚蘭提出了一個對鑰匙與鎖模型進行修正的理論:酶的結構相對靈活,底物與酶(活性位點)作用時,活性位點會不斷改變結構[33]。底物不是簡單地與一個剛性的活性位點結合。組成活性位點的氨基酸側鏈的準確有序排布保證酶能執行催化功能。糖苷酶等酶,當底物分子與活性位點結合時,底物分子亦會發生輕微的形狀改變[34]。直到底物與酶發生完全結合,分子形狀和電荷排佈都最終確定,活性位點都會不斷發生結構變化[35]誘導契合可以通過結構校對(conformational proofreading)機制在噪音和競爭物存在的條件下增強分子識別的保真度[36]。
催化机理
酶可以通過多種方式加快化學反應的進行速度,基本機理都是降低反應的活化能(ΔG‡,吉布斯自由能)[37]。
- 穩定過度態:
- 產生一個與過度態所帶電荷互補的環境,以降低其能量[38]。
- 提供不同的反應途徑:
- 先和底物發生初步反應,與底物形成共價鍵连接,产生一個能量較低的中間态[39]。
- 降低底物基態的穩定性:
- 结合底物后引起底物扭曲,从而降低底物基态与過渡態之间的能差[40]。
- 通過改變底物的排列方式,減少反應的熵變[41],此機制對催化的貢獻較小。[42]。
酶可以同时使用以上多種催化机制来催化反應。比如,胰蛋白酶先通過一個催化三聯體进行共价催化产生中间态,再借助氧負離子洞(oxyanion hole)稳定過渡態的電荷排佈,水解過程的完成則依賴有序排列的水分子底物。
动态结构
酶的結構並不是剛性的、恆定不變的。酶的內部結構會發生複雜的動力學變化。酶的結構可能在反應過程中發生變化,單個氨基酸殘基、一個轉角、一個二級結構單位,乃至整個結構域的位置都可能發生改變。酶內部結構的變化能使差異度不大、且能發生互變的多种构象体在熱力學平衡狀態下共存,从而形成所谓构象系综(conformational ensemble)。在系综狀態下,酶的每一結構狀態或者构象体都可能與酶的功能的某一部分有關。舉例來說,二氢叶酸还原酶的不同構象就分別和底物結合、催化、輔助因子釋放、產物釋放相關[43]。
别构调节
別構中心是酶结构上的結合口袋,远离活性中心,可以与细胞环境中的一些分子结合。和別構中心結合的分子往往可以改變酶的構象或是酶的动态结构,从而影響酶活性中心的反應速率[44]。因此,別構反應可以使酶被激活或被抑制。別構调节对代謝通路非常重要,代謝酶往往可以和通路上游或是下游的代謝物发生別構效应,而产生反馈调控,即根据通路其他部分上的代谢通量來調整酶的活性[45]。
輔助因子

轉酮酶(Transketolase)以及硫胺素焦磷酸的結構。黃色部分代表硫胺素焦磷酸,黑色部分代表木酮糖-5-磷酸(PDB 4KXV)
一些酶並不需要額外的組分就能就能正常發揮作用,另外一些酶則要在和輔助因子(cofactor)結合後才能顯示出活性[46]。輔助因子可以是無機物(如金屬離子、铁硫簇),也可以是有機物(比如黃素和血紅素)。有機輔助
因子如果在反應中會與酶分離則爲輔酶(coenzyme),如果與酶緊密結合則爲輔基(prosthetic groups)。有機的輔基可能與酶發生共價結合(丙酮酸羧化酶與生物素(biotin)之間即發生共價結合)[47]。
碳酸酐酶(carbonic anhydrase)即是一類含有輔助因子的酶。理查德森圖(ribbon diagram)顯示,碳酸酐酶的肽鏈環繞在一個鋅離子(值得注意的是,鋅離子亦是活性位點的一部分)周圍,並與這個鋅離子結合在一起[48]這些與酶緊密結合的離子或分子通常位於活性位點之中,並且能夠參與催化反應[1]:8.1.1。例如,黃素以及血紅素即會參與氧化還原反應[1]:17。
需要輔助因子才可以發揮作用的酶,處於未與輔助因子狀態時稱爲「脫輔酶」(apoenzymes或apoproteins),當其與輔助因子結合後則稱爲「全酶」(holoenzyme)。不過,值得注意的是,名詞全酶亦可以指含有多個亞基的酶,如DNA聚合酶。不過,在本文中,全酶是指含有所有發揮活性所需的亞基的酶[1]:8.1.1。
輔酶
輔酶是一類與酶結合的小分子有機物,輔酶與酶結合的強度因輔酶和酶的種類而異,或強或弱。輔酶能夠將化學基團從一個酶轉移到另一個酶上[49]。NADH、NADPH爲兩種常見的輔酶。核黃素、硫胺、葉酸等維生素類輔酶人體不能合成,需要通過膳食補充。輔酶能攜帶化學基團,如NAD或NADP能攜帶氫離子(H+)、ATP能攜帶磷酸基團、輔酶A能攜帶乙酰基團、葉酸基團能攜帶甲酰基、次甲基,或甲基、S-腺苷甲硫氨酸能攜帶甲基[49]。
輔酶在酶促反應發生後化學結構會發生改變。因此,可以將看作一類普遍存在的特殊底物。許多酶都有與之匹配的輔酶,比如,已知的酶中有大約1000種酶使用NADH作爲輔酶(2015年的數據)[50]。
輔酶通常能不斷再生,濃度能始終維持在一個恆定不變的水平上。舉例來說,NADPH能通過磷酸戊糖途径再生,S-腺苷甲硫氨酸能通過甲硫氨酸腺苷轉移酶催化的反應生成。持續不斷的再生意味着總量不多的輔酶也能以很快的速度消耗。舉例來說,人類每天會更新重量與自身體重相等的ATP[51]。
热力学

有酶或无酶催化反应体系中反应进程与能量关系图示。可以看出,当反应没有酶的催化时,底物通常需要获得较高的活化能才能到达过渡态,然后才能生成产物;而当反应体系中有酶催化时,通过酶对过渡态的稳定作用,降低了达到过渡态所需能量,从而降低了整个反应所需的能量。
与其他催化剂一样,酶并不改变反应的平衡常数,而是通过降低反应的活化能来加快反应速率(见右图)。通常情况下,反应在酶存在或不存在的两种条件下,其反应方向是相同的,只是前者的反应速度更快一些。但必须指出的是,在酶不存在的情况下,底物可以通过其他不受催化的“自由”反应生成不同的产物,原因是这些不同产物的形成速度更快。
酶可以连接两个或多个反应,因此可以用一个热力学上更容易发生的反应去“驱动”另一个热力学上不容易发生的反应。例如,细胞常常通过ATP被酶水解所产生的能量来驱动其他化学反应。[52]
酶可以同等地催化正向反应和逆向反应,而并不改变反应自身的化学平衡。例如,碳酸酐酶可以催化如下两个互逆反应,催化哪一种反应则是依赖于反应物浓度。[53]
CO2+H2O∗→H2CO3displaystyle mathrm CO_2+H_2O^mathrm quad * !!!!!!!!overrightarrow qquad H_2CO_3(组织内;高CO2浓度下)
H2CO3∗→CO2+H2Odisplaystyle mathrm H_2CO_3^mathrm quad * !!!!!!!!overrightarrow qquad CO_2+H_2O(肺中;低CO2浓度下)
反应式中“*”表示“碳酸酐酶”
当然,如果反应平衡极大地趋向于某一方向,比如释放高能量的反应,而逆反应不可能有效的发生,则此时酶实际上只催化热力学上允许的方向,而不催化其逆反应。
动力学

单一底物的酶催化反应机理:酶(表示为“E”)结合底物(表示为“S”),并通过催化反应生成产物(表示为“P”)。
未解決的化學問題:为什么一些酶的表现快于扩散动力学? |
酶动力学是研究酶结合底物能力和催化反应速率的科学。研究者通过酶反应分析法(enzyme assay)来获得用于酶动力学分析的反应速率数据。
1902年,维克多·亨利提出了酶动力学的定量理论;[54]随后该理论得到他人证实并扩展为米氏方程。[55]亨利最大贡献在于其首次提出酶催化反应由两步组成:首先,底物可逆地结合到酶上,形成酶-底物复合物;然后,酶完成对对应化学反应的催化,并释放生成的产物(见左图)。
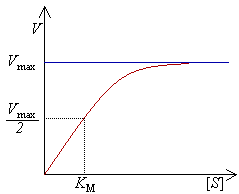
酶初始反应速率(表示为“V”)与底物浓度(表示为“[S]”)的关系曲线。随着底物浓度不断提高,酶的反应速率也趋向于最大反应速率(表示为“Vmax”)。
酶可以在一秒钟内催化数百万个反应。例如,乳清酸核苷5'-磷酸脱羧酶所催化的反应在无酶情况下,需要七千八百万年才能将一半的底物转化为产物;而同样的反应过程,如果加入这种脱羧酶,则需要的时间只有25毫秒。[56]酶催化速率依赖于反应条件和底物浓度。如果反应条件中存在能够将蛋白解链的因素,如高温、极端的pH和高的盐浓度,都会破坏酶的活性;而提高反应体系中的底物浓度则会增加酶的活性。在酶浓度固定的情况下,随着底物浓度的不断升高,酶催化的反应速率也不断加快并趋向于最大反应速率(Vmax,见右图的饱和曲线)。出现这种现象的原因是,当反应体系中底物的浓度升高,越来越多自由状态下的酶分子结合底物形成酶-底物复合物;当所有酶分子的活性位点都被底物饱和结合,即所有酶分子形成酶-底物复合物时,催化的反应速率达到最大。当然,Vmax并不是酶唯一的动力学常数,要达到一定反应速率所需的底物浓度也是一个重要的动力学指标。这一动力学指标即米氏常数(Km),指的是达到Vmax值一半的反应速率所需的底物浓度(见右图)。对于特定的底物,每一种酶都有其特征Km值,表示底物与酶之间的结合强度(Km值越低,结合越牢固,亲和力越高)。另一个重要的动力学指标是kcat,定义为一个酶活性位点在一秒钟内催化底物的数量,用于表示酶催化特定底物的能力。
酶的催化效率可以用kcat/Km来衡量。这一表示式又被称为特异性常数,其包含了催化反应中所有步骤的反应常数。由于特异性常数同时反映了酶对底物的亲和力和催化能力,因此可以用于比较不同酶对于特定底物的
催化效率或同一种酶对于不同底物的催化效率。特异性常数的理论最大值,又称为扩散极限,约为108至109 M−1s−1;此时,酶与底物的每一次碰撞都会导致底物被催化,因此产物的生成速率不再为反应速率所主导,而分子的扩散速率起到了决定性作用。酶的这种特性被称为“催化完美性”或“动力学完美性”。相关的酶的例子有磷酸丙糖异构酶、碳酸酐酶、乙酰胆碱酯酶、过氧化氢酶、延胡索酸酶、β-内酰胺酶和超氧化物歧化酶。
米氏方程是基于质量作用定律而确立的,而该定律则基于自由扩散和热动力学驱动的碰撞这些假定。然而,由于酶/底物/产物的高浓度和相分离或者一维/二维分子运动,许多生化或细胞进程明显偏离质量作用定律的假定。[57]在这些情况下,可以应用分形米氏方程。[58][59][60][61]
存在一些酶,它们的催化产物动力学速率甚至高于分子扩散速率,这种现象无法用目前公认的理论来解释。有多种理论模型被提出来解释这类现象。其中,部分情况可以用酶对底物的附加效应来解释,即一些酶被认为可以通过双偶极电场来捕捉底物以及将底物以正确方位摆放到催化活性位点。另一种理论模型引入了基于量子理论的穿隧效应,即质子或电子可以穿过激活能垒(就如同穿过隧道一般),但关于穿隧效应还有较多争议。[62][63]有报道发现色胺中质子存在量子穿隧效应。[64]因此,有研究者相信在酶催化中也存在着穿隧效应,可以直接穿过反应能垒,而不是像传统理论模型的方式通过降低能垒达到催化效果。有相关的实验报道提出在一种醇脱氢酶的催化反应中存在穿隧效应,[65]但穿隧效应是否在酶催化反应中普遍存在并未有定论。[66]
抑制作用
酶的催化活性可以被多种抑制剂所降低。[67]

不同的抑制类型。分类参考自[68]。图中,“E”表示酶;“I”表示抑制剂;“S”表示底物;“P”表示产物。
可逆抑制作用
可逆抑制作用的类型有多种,它们的共同特点在于抑制剂对酶活性的抑制反应具有可逆性。
竞争性抑制作用
抑制剂与底物竞争结合酶的活性位点(抑制剂和底物不能同时结合到活性位点),也就意味着它们不能同时结合到酶上。[69]对于竞争性抑制作用,催化反应的最大反应速率值没有变,但是需要更高的底物浓度,反映在表观Km值的增加。
非竞争性抑制作用
非竞争性抑制抑制剂可以与底物同时结合到酶上,即抑制剂不结合到活性位点。酶-抑制剂复合物(EI)或酶-抑制剂-底物复合物(EIS)都没有催化活性。与竞争性抑制作用相比,非竞争性抑制作用不能通过提高底物浓度来达到所需反应速度,即表观最大反应速率Vmax的值变小;而同时,由于抑制剂不影响底物与酶的结合,因此Km值保持不变。
反竞争性抑制作用
反竞争性抑制作用比较少见:抑制剂不能与处于自由状态下的酶结合,而只能和酶-底物复合物(ES)结合,在酶反应动力学上表现为Vmax和Km值都变小。这种抑制作用可能发生在多亚基酶中。
复合抑制作用
这种抑制作用与非竞争性抑制作用比较相似,区别在于EIS复合物残留有部分酶的活性。在许多生物体中,这类抑制剂可以作为负反馈机制的组成部分。若一个酶体系生产了过多的产物,那么产物就会抑制合成该产物的酶体系中第一个酶的活性,这就可以保证一旦合成足够多的产物后,该产物的合成速率会下降或停止。受这种抑制作用调控的酶通常为多亚基酶,并具有与调控产物结合的别构结合位点。这种抑制作用的反应速率与底物浓度的关系图不再是双曲线形而是S形。
不可逆抑制作用
不可逆抑制剂可以与酶结合形成共价连接,而其他抑制作用中酶与抑制剂之间都是非共价结合。这种抑制作用是不可逆的,酶一旦被抑制后就无法再恢复活性状态。这类抑制剂包括二氟甲基鸟氨酸(一种可用于治疗寄生虫导致的昏睡症的药物[70])、苯甲基磺酰氟(PMSF)、青霉素和阿司匹林。这些药物都是与酶活性位点结合并被激活,然后与活性位点处的一个或多个氨基酸残基发生不可逆的反应形成共价连接。
抑制剂的用途
酶抑制剂常被用作药物,同样也可以被作为毒药使用。而药物和毒药之间的差别通常非常小,大多数的药物都有一定程度的毒性,正如帕拉塞尔苏斯所言:“所有东西都有毒,没有什么是无毒的”(“In all things there is a poison, and there is nothing without a poison”)。[71]相同的,抗生素和其他抗感染药物只是特异性地对病原体而不是对宿主有毒性。
一个获得广泛应用的抑制剂药物是阿司匹林,它可以抑制环加氧酶的活性,而环加氧酶可以生产炎症反应信使前列腺素,因此,阿司匹林可以起到抑制疼痛与炎症的作用。而剧毒毒药氰化物可以通过结合细胞色素氧化酶位点处的铜和铁原子不可逆地抑制酶活性,从而抑制细胞的呼吸作用。[72]
生物学功能
在生物体内,酶发挥着非常广泛的功能。信号转导和细胞活动的调控都离不开酶,特别是激酶和磷酸酶的参与。[73]酶也能产生运动,通过催化肌球蛋白上ATP的水解产生肌肉收缩,并且能够作为细胞骨架的一部分参与运送胞内物质。[74]一些位于细胞膜上的ATP酶作为离子泵参与主动运输。一些生物体中比较奇特的功能也有酶的参与,例如荧光素酶可以为萤火虫发光。[75]病毒中也含有酶,或参与侵染细胞(如HIV整合酶和逆转录酶),或参与病毒颗粒从宿主细胞的释放(如流感病毒的神经氨酸酶)。
酶的一个非常重要的功能是参与在动物消化系统的工作。以淀粉酶和蛋白酶为代表的一些酶可以将进入消化道的大分子(淀粉和蛋白质)降解为小分子,以便于肠道吸收。淀粉不能被肠道直接吸收,而酶可以将淀粉水解为麦芽糖或更进一步水解为葡萄糖等肠道可以吸收的小分子。不同的酶分解不同的食物底物。在草食性反刍动物的消化系统中存在一些可以产生纤维素酶的细菌,纤维素酶可以分解植物细胞壁中的纤维素,从而提供可被吸收的养料。[76]
代谢

糖酵解酶及其在糖酵解的代谢途径的功能
多个酶以某一特定的顺序发挥功能,共同构成了代谢途径。在代谢途径中,前一个酶的产物是后一个酶的底物;每个酶催化反应后,产物被传递到另一个酶。有些情况下,不同的酶可以平行地催化同一个反应,从而允许进行更为复杂的调控:比如一个酶可以以较低的活性持续地催化该反应,而另一个酶在被诱导后可以较高的活性进行催化。
酶的存在确定了整个代谢按正确的途径进行;而一旦没有酶的存在,代谢既不能按所需步骤进行,也无法以足够的速度完成合成以满足细胞的需要。实际上如果没有酶,代谢途径,如糖酵解,无法独立进行。例如,葡萄糖可以直接与ATP反应使得其一个或多个碳原子被磷酸化;在没有酶的催化时,这个反应进行得非常缓慢以致可以忽略;而一旦加入己糖激酶,在6位上的碳原子的磷酸化反应获得极大加速,虽然其他碳原子的磷酸化反应也在缓慢进行,但在一段时间后检测可以发现,绝大多数产物为葡萄糖-6-磷酸。[77]于是每个细胞就可以通过这样一套功能性酶来完成代谢途径的整个反应网络。
活性控制
细胞内有五种控制酶催化活性的机制:
- 根据外界环境的变化,细胞可以增强或减弱酶的生产(即酶相关基因的转录和翻译)。这属于一种基因调控,被称为酶的诱导和抑制。例如,当环境中出现如青霉素这样的抗生素时,部分细菌可以对抗生素产生抗性,其原因就在于细菌体内的β-半乳糖苷酶被诱导而大量生产,这种酶可以水解青霉素分子上关键的β-乳胺环。另一个例子是在人体肝脏中存在一类酶对于药物代谢非常重要的酶,细胞色素P450氧化酶;对这一类酶的诱导或抑制,会导致藥物相互作用。
- 通过将特定的酶分隔在特定的细胞组分中,细胞可以完成不同的代谢途径。例如,脂肪酸的合成是由细胞溶质、内质网和高尔基体中的一系列酶所完成,而脂肪酸的降解(以提供能量)是在线粒体中由另一系列酶通过β-氧化来完成。[78]
- 酶可以被抑制剂与激活剂所调控。例如,一个代谢途径中的终产物常常是这一途径中第一个酶的抑制剂,从而调控这一代谢途径的产物量。这种调控机制被称为负反馈机制,因为终产物的合成量是受其自身浓度调控。负反馈机制可以根据细胞的需要,有效地调节中间代谢物的合成速率,从而使细胞的能量和物质的分配更为高效,并防止多余产物的合成。控制酶的作用,可以在生物体内维持一个稳定的内部环境(即体内平衡)。
翻译后修饰也可以调控酶的活性。这些修饰包括磷酸化、肉豆蔻酸化和糖基化。例如,细胞接受胰岛素信号后,对包括糖原合酶在内的多个酶进行磷酸化,帮助控制糖原的合成或降解,使得细胞可以对血糖的变化产生反应。[79]另一个翻译后修饰的例子是多肽链的剪切。胰凝乳蛋白酶,一种消化性蛋白酶,是产生于胰脏中的无活性的胰凝乳蛋白酶原,这一蛋白通过运输到达胃后才被激活。这种方式有效地防止了胰凝乳蛋白酶在进入肠之前消化胰脏或其他组织。这种无活性的酶的前体被命名为酶原。- 还有一些酶可以通过定位到不同环境后而被激活,比如从还原态的环境(细胞质)到氧化态环境(细胞周质空间),从高pH环境到低pH环境等。流感病毒的红血球凝集素蛋白就是一个例子:当它接触到宿主细胞囊泡的酸性环境时,它的构象立刻发生变化,导致其获得激活。[80]
相关疾病
苯丙氨酸羟化酶. 由PDB 1KW0生成。
酶的活性必须严格控制以维持体内平衡,对于能够影响一个关键酶的功能的任何基因缺陷(如突变导致活性变化,过量表达、过低表达或删除突变)都可能导致遗传性疾病发生。许多事实显示,一种致命疾病的病因可以只是由于人体中的数千种酶中的一种发生功能故障。
苯丙酮尿症:此種病症是典型的酶相关病例之一。病因是苯丙氨酸羟化酶(其功能是催化苯丙氨酸降解过程中的第一步)上一个氨基酸位点发生了突变,导致体内苯丙氨酸和相关产物的水平过高,如果没有得到合适的治疗,会进一步导致智能障碍。[81]
卟啉病:该病是由于血红素生物合成途径中特定酶的酶活性过低(基因突变或其他原因导致),使得中间产物卟啉的产生和排泄异常,在一定诱因(如阳光照射)下,可导致皮肤或其他组织器官发生病变。[82]- 当生殖细胞中编码DNA修复相关酶的基因发生突变,其结果会导致遗传性癌症综合病征,如着色性干皮症。[83]DNA修复酶的缺陷导致人体丧失修复突变基因的能力。发生的突变不断积累,最终使得患者有多种癌症发生。
酶的口服给药,可用于治疗多种疾病(如胰腺功能不全和乳糖不耐受症)。由于酶作为蛋白质可能在消化道环境中失活或被降解,因此一种非侵入性的成像方法被开发用于监测作为药物的酶在消化道中的活性变化。[84]
工业應用
酶因爲能高效催化特定反應,已在化工等行業得到廣泛應用。總的來說,酶的應用因爲它們能催化的反應數目少、在有機溶劑中以及高溫環境下不穩定而受到限制。因此,酶工程這一熱門學科應運而生。酶工程旨在藉助合理的設計或體外進化的方法研發具有新特性的酶[85][86]。目前,酶工程學已取得了一定成果,研究人員甚至已「從頭」(即不以任何自然界中的酶爲模板)設計出了一些能催化在自然界中不能發生的反應的酶[87]。
| 應用領域 | 酶 | 用途 |
|---|---|---|
生物燃料工業 | 纖維素酶 | 將纖維素分解可通過發酵轉化爲纖維素乙醇的糖[88] |
木質酶 | 對準備用於生物燃料生產的生物質進行預處理[88] | |
生物洗滌劑 | 蛋白酶、澱粉酶、脂肪酶 | 洗衣或清洗餐具時去除蛋白質、澱粉、脂肪或油漬[89] |
甘露聚糖酶 | 去除食物食品污渍[89] | |
酿酒業 | 淀粉酶、葡聚糖酶、蛋白酶 | 從麦芽中分解多醣及蛋白質[90]:150–9 |
β-葡聚醣酶 | 提高麦汁和啤酒过滤特征[90]:545 | |
淀粉葡糖苷酶及支链淀粉酶 | 製作低熱量啤酒及调整发酵特性[90]:575 | |
乙酰乳酸脫羧酶(ALDC) | 利用減少丁二酮形成來提昇发酵效率[91] | |
烹饪用 | 木瓜蛋白酶 | 使肉變嫰,容易烹饪[92] |
乳品業 | 凝乳酶 | 在奶酪生产過程中酸化蛋白质[93] |
脂酶 | 製作卡芒贝尔奶酪及像罗克福干酪之類的藍乾酪[94] | |
食品加工 | 淀粉酶 | 從淀粉製造糖類,例如製作高果糖玉米糖浆[95] |
蛋白酶 | 降低面粉中的蛋白質比例,例如用在餅乾製造中[96] | |
胰蛋白酶 | 製作防过敏的嬰兒食品[96] | |
纤维素酶、果胶酶 | 澄清果汁[97] | |
分子生物學 | 核酸酶、聚合酶、DNA連接酶 | 藉助限制性核酸內切酶以及PCR技術產生重組DNA[1]:6.2 |
造紙業 | 木聚糖酶、半纤维素酶及木质素过氧化物酶 | 從木浆中移除木质素[98] |
个人护理 | 蛋白酶 | 清除隱形眼鏡上的蛋白質,防止感染。[99] |
澱粉工業 | 淀粉酶 | 將淀粉轉換成葡萄糖及各種糖漿[100] |
參見
- 酶列表
- 酶數據庫
- BRENDA
- ExPASy
- IntEnz
- KEGG
- MetaCyc
參考
^ 1.01.11.21.31.41.51.61.71.81.9 Stryer L, Berg JM, Tymoczko JL. Biochemistry 5th. San Francisco: W.H. Freeman. 2002. ISBN 0-7167-4955-6.
^ Schomburg I, Chang A, Placzek S, Söhngen C, Rother M, Lang M, Munaretto C, Ulas S, Stelzer M, Grote A, Scheer M, Schomburg D. BRENDA in 2013: integrated reactions, kinetic data, enzyme function data, improved disease classification: new options and contents in BRENDA. Nucleic Acids Research. January 2013, 41 (Database issue): D764–72. PMC 3531171. PMID 23203881. doi:10.1093/nar/gks1049.
^ Radzicka A, Wolfenden R. A proficient enzyme. Science. January 1995, 267 (5194): 90–931. Bibcode:1995Sci...267...90R. PMID 7809611. doi:10.1126/science.7809611.
^ Callahan BP, Miller BG. OMP decarboxylase—An enigma persists. Bioorganic Chemistry. December 2007, 35 (6): 465–9. PMID 17889251. doi:10.1016/j.bioorg.2007.07.004.
^ (法文)de Réaumur, RAF. Observations sur la digestion des oiseaux. Histoire de l'academie royale des sciences. 1752, 1752: 266, 461.
^ (英文)Williams, H. S.(1904)A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences Harper and Brothers (New York) Accessed 04 April 2007
^ (英文)Dubos J. Louis Pasteur: Free Lance of Science, Gollancz. Quoted in Manchester K. L.(1995)Louis Pasteur(1822–1895)—chance and the prepared mind.. Trends Biotechnol. 1951, 13 (12): 511–515. PMID 8595136.
^ (英文)诺贝尔奖获得者爱德华·比希纳的简历Accessed 04 April 2007
^ (英文)爱德华·比希纳在1907年的诺贝尔奖获奖演说Accessed 04 April 2007
^ (英文)1946年度诺贝尔化学奖获得者Accessed 04 April 2007
^ (英文)Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Structure of hen egg-white lysozyme. A three-dimensional Fourier synthesis at 2 Angstrom resolution.. Nature. 1965, 22 (206): 757–761. PMID 5891407.
^ (英文)1989年度诺贝尔化学奖授予了托马斯·切赫和悉尼·奥尔特曼以奖励他们发现RNA分子的催化性质。
^ Nomenclature Committee. Classification and Nomenclature of Enzymes by the Reactions they Catalyse. International Union of Biochemistry and Molecular Biology (NC-IUBMB). School of Biological and Chemical Sciences, Queen Mary, University of London.
^ Nomenclature Committee. EC 2.7.1.1. International Union of Biochemistry and Molecular Biology (NC-IUBMB). School of Biological and Chemical Sciences, Queen Mary, University of London.
^ Anfinsen CB. Principles that govern the folding of protein chains. Science. July 1973, 181 (4096): 223–30. Bibcode:1973Sci...181..223A. PMID 4124164. doi:10.1126/science.181.4096.223.
^ Dunaway-Mariano D. Enzyme function discovery. Structure (London, England : 1993). November 2008, 16 (11): 1599–600. PMID 19000810. doi:10.1016/j.str.2008.10.001.
^ Petsko GA, Ringe D. Chapter 1: From sequence to structure. Protein structure and function. London: New Science. 2003: 27. ISBN 978-1405119221.
^ Chen LH, Kenyon GL, Curtin F, Harayama S, Bembenek ME, Hajipour G, Whitman CP. 4-Oxalocrotonate tautomerase, an enzyme composed of 62 amino acid residues per monomer. The Journal of Biological Chemistry. September 1992, 267 (25): 17716–21. PMID 1339435.
^ Smith S. The animal fatty acid synthase: one gene, one polypeptide, seven enzymes. FASEB Journal. December 1994, 8 (15): 1248–59. PMID 8001737.
^ The Catalytic Site Atlas. The European Bioinformatics Institute. [4 April 2007].
^ 21.021.1 Suzuki H. Chapter 7: Active Site Structure. How Enzymes Work: From Structure to Function. Boca Raton, FL: CRC Press. 2015: 117–140. ISBN 978-981-4463-92-8.
^ Krauss G. The Regulations of Enzyme Activity. Biochemistry of Signal Transduction and Regulation 3rd. Weinheim: Wiley-VCH. 2003: 89–114. ISBN 9783527605767.
^ Jocelyn E.KREBS; 等. Gene XI. JONES&BARTLETT LEARNING(高等教育出版社出版). 2014. ISBN 978-7-04-039649-2. 引文格式1维护:显式使用等标签 (link)
^ Jaeger KE, Eggert T. Enantioselective biocatalysis optimized by directed evolution. Current Opinion in Biotechnology. August 2004, 15 (4): 305–13. PMID 15358000. doi:10.1016/j.copbio.2004.06.007.
^ Shevelev IV, Hübscher U. The 3' 5' exonucleases. Nature Reviews Molecular Cell Biology. May 2002, 3 (5): 364–76. PMID 11988770. doi:10.1038/nrm804.
^ Ibba M, Soll D. Aminoacyl-tRNA synthesis. Annual Review of Biochemistry. 2000, 69: 617–50. PMID 10966471. doi:10.1146/annurev.biochem.69.1.617.
^ Rodnina MV, Wintermeyer W. Fidelity of aminoacyl-tRNA selection on the ribosome: kinetic and structural mechanisms. Annual Review of Biochemistry. 2001, 70: 415–35. PMID 11395413. doi:10.1146/annurev.biochem.70.1.415.
^ Zenkin N, Yuzenkova Y, Severinov K. Transcript-assisted transcriptional proofreading. Science. July 2006, 313 (5786): 518–20. Bibcode:2006Sci...313..518Z. PMID 16873663. doi:10.1126/science.1127422.
^ Khersonsky O, Tawfik DS. Enzyme promiscuity: a mechanistic and evolutionary perspective. Annual Review of Biochemistry. 2010, 79: 471–505. PMID 20235827. doi:10.1146/annurev-biochem-030409-143718.
^ O'Brien PJ, Herschlag D. Catalytic promiscuity and the evolution of new enzymatic activities. Chemistry & Biology. April 1999, 6 (4): R91–R105. PMID 10099128. doi:10.1016/S1074-5521(99)80033-7.
^ Fischer E. Einfluss der Configuration auf die Wirkung der Enzyme [Influence of configuration on the action of enzymes]. Berichte der Deutschen chemischen Gesellschaft zu Berlin. 1894, 27 (3): 2985–93. doi:10.1002/cber.18940270364 (德语). From page 2992: "Um ein Bild zu gebrauchen, will ich sagen, dass Enzym und Glucosid wie Schloss und Schlüssel zu einander passen müssen, um eine chemische Wirkung auf einander ausüben zu können." (To use an image, I will say that an enzyme and a glucoside [i.e., glucose derivative] must fit like a lock and key, in order to be able to exert a chemical effect on each other.)
^ Cooper GM. Chapter 2.2: The Central Role of Enzymes as Biological Catalysts. The Cell: a Molecular Approach 2nd. Washington (DC ): ASM Press. 2000. ISBN 0-87893-106-6.
^ Koshland DE. Application of a Theory of Enzyme Specificity to Protein Synthesis. Proceedings of the National Academy of Sciences of the United States of America. February 1958, 44 (2): 98–104. Bibcode:1958PNAS...44...98K. PMC 335371. PMID 16590179. doi:10.1073/pnas.44.2.98.
^ Vasella A, Davies GJ, Böhm M. Glycosidase mechanisms. Current Opinion in Chemical Biology. October 2002, 6 (5): 619–29. PMID 12413546. doi:10.1016/S1367-5931(02)00380-0.
^ Boyer R. Chapter 6: Enzymes I, Reactions, Kinetics, and Inhibition. Concepts in Biochemistry 2nd. New York, Chichester, Weinheim, Brisbane, Singapore, Toronto.: John Wiley & Sons, Inc. 2002: 137–8. ISBN 0-470-00379-0. OCLC 51720783.
^ Savir Y, Tlusty T. Scalas E, 编. Conformational proofreading: the impact of conformational changes on the specificity of molecular recognition (PDF). PLoS ONE. 2007, 2 (5): e468. Bibcode:2007PLoSO...2..468S. PMC 1868595. PMID 17520027. doi:10.1371/journal.pone.0000468.
^ Fersht A. Enzyme Structure and Mechanism. San Francisco: W.H. Freeman. 1985: 50–2. ISBN 0-7167-1615-1.
^ Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH. Electrostatic basis for enzyme catalysis. Chemical Reviews. August 2006, 106 (8): 3210–35. PMID 16895325. doi:10.1021/cr0503106.
^ Cox MM, Nelson DL. Chapter 6.2: How enzymes work. Lehninger Principles of Biochemistry 6th. New York, N.Y.: W.H. Freeman. 2013: 195. ISBN 978-1464109621.
^ Benkovic SJ, Hammes-Schiffer S. A perspective on enzyme catalysis. Science. August 2003, 301 (5637): 1196–202. Bibcode:2003Sci...301.1196B. PMID 12947189. doi:10.1126/science.1085515.
^ Jencks WP. Catalysis in Chemistry and Enzymology. Mineola, N.Y: Dover. 1987. ISBN 0-486-65460-5.
^ Villa J, Strajbl M, Glennon TM, Sham YY, Chu ZT, Warshel A. How important are entropic contributions to enzyme catalysis?. Proceedings of the National Academy of Sciences of the United States of America. October 2000, 97 (22): 11899–904. Bibcode:2000PNAS...9711899V. PMC 17266. PMID 11050223. doi:10.1073/pnas.97.22.11899.
^ Ramanathan A, Savol A, Burger V, Chennubhotla CS, Agarwal PK. Protein conformational populations and functionally relevant substates. Acc. Chem. Res. 2014, 47 (1): 149–56. PMID 23988159. doi:10.1021/ar400084s.
^ Tsai CJ, Del Sol A, Nussinov R. Protein allostery, signal transmission and dynamics: a classification scheme of allosteric mechanisms. Mol Biosyst. 2009, 5 (3): 207–16. PMC 2898650. PMID 19225609. doi:10.1039/b819720b.
^ Changeux JP, Edelstein SJ. Allosteric mechanisms of signal transduction. Science. June 2005, 308 (5727): 1424–8. Bibcode:2005Sci...308.1424C. PMID 15933191. doi:10.1126/science.1108595.
^ de Bolster MW. Glossary of Terms Used in Bioinorganic Chemistry: Cofactor. International Union of Pure and Applied Chemistry. 1997 [30 October 2007].
^ Chapman-Smith A, Cronan JE. The enzymatic biotinylation of proteins: a post-translational modification of exceptional specificity. Trends Biochem. Sci. 1999, 24 (9): 359–63. PMID 10470036. doi:10.1016/s0968-0004(99)01438-3.
^ Fisher Z, Hernandez Prada JA, Tu C, Duda D, Yoshioka C, An H, Govindasamy L, Silverman DN, McKenna R. Structural and kinetic characterization of active-site histidine as a proton shuttle in catalysis by human carbonic anhydrase II. Biochemistry. February 2005, 44 (4): 1097–115. PMID 15667203. doi:10.1021/bi0480279.
^ 49.049.1 Wagner AL. Vitamins and Coenzymes. Krieger Pub Co. 1975. ISBN 0-88275-258-8.
^ BRENDA The Comprehensive Enzyme Information System. Technische Universität Braunschweig. [23 February 2015].
^ Törnroth-Horsefield S, Neutze R. Opening and closing the metabolite gate. Proceedings of the National Academy of Sciences of the United States of America. December 2008, 105 (50): 19565–6. Bibcode:2008PNAS..10519565T. PMC 2604989. PMID 19073922. doi:10.1073/pnas.0810654106.
^ (英文)Ferguson, S. J.; Nicholls, David; Ferguson, Stuart. Bioenergetics 3 3rd. San Diego: Academic. 2002. ISBN 0-12-518121-3.
^ (英文)Maren TH. Carbonic anhydrase: chemistry, physiology, and inhibition. Physiol Rev. 1967, 47 (4): 595–781. PMID 4964060.
^ (法文)Henri,V. Theorie generale de l'action de quelques diastases. Compt. rend. hebd. Acad. Sci. Paris. 1902, 135: 916–919.
^ (德文)Michaelis L., Menten M. Die Kinetik der Invertinwirkung. Biochem. Z. 1913, 49: 333–369. English translation Accessed 6 April 2007
^ (英文)Radzicka A, Wolfenden R. A proficient enzyme.. Science. 1995, 6 (267): 90–931. PMID 7809611.
^ (英文)Ellis RJ. Macromolecular crowding: obvious but underappreciated. Trends Biochem. Sci. 2001, 26 (10): 597–604. PMID 11590012.
^ (英文)Kopelman R. Fractal Reaction Kinetics. Science. 1988, 241 (4873): 1620–26. doi:10.1126/science.241.4873.1620.
^ (英文)Savageau MA. Michaelis-Menten mechanism reconsidered: implications of fractal kinetics. J. Theor. Biol. 1995, 176 (1): 115–24. PMID 7475096.
^ (英文)Schnell S, Turner TE. Reaction kinetics in intracellular environments with macromolecular crowding: simulations and rate laws. Prog. Biophys. Mol. Biol. 2004, 85 (2–3): 235–60. PMID 15142746.
^ (英文)Xu F, Ding H. A new kinetic model for heterogeneous (or spatially confined) enzymatic catalysis: Contributions from the fractal and jamming (overcrowding) effects. Appl. Catal. A: Gen. 2007, 317 (1): 70–81. doi:10.1016/j.apcata.2006.10.014.
^ (英文)Garcia-Viloca M., Gao J., Karplus M., Truhlar D. G. How enzymes work: analysis by modern rate theory and computer simulations.. Science. 2004, 303 (5655): 186–195. PMID 14716003.
^ (英文)Olsson M. H., Siegbahn P. E., Warshel A. Simulations of the large kinetic isotope effect and the temperature dependence of the hydrogen atom transfer in lipoxygenase. J. Am. Chem. Soc. 2004, 126 (9): 2820–1828. PMID 14995199.
^ (英文)Masgrau L., Roujeinikova A., Johannissen L. O., Hothi P., Basran J., Ranaghan K. E., Mulholland A. J., Sutcliffe M. J., Scrutton N. S., Leys D. Atomic Description of an Enzyme Reaction Dominated by Proton Tunneling. Science. 2006, 312 (5771): 237–241. PMID 16614214.
^ (英文)Kohen, A., Cannio, R., Bartolucci, S., Klinman, J. P. Enzyme dynamics and hydrogen tunnelling in a thermophilic alcohol dehydrogenase. Nature. 1999, 399 (6735): 496–9. PMID 10365965.
^ (英文)Ball, P. Enzymes: by chance, or by design?. Nature. 2004, 431 (7007): 396–7. PMID 15385982.
^ Laurence A. Moran, Robert Horton, Gray Scrimgeour & Marc D. Perry. Properties of Enzymes. Principles of Biochemistry (Fifth Edition) [生物化学原理]. 培生出版集團. 2011: 148–152. ISBN 0-321-70733-8.
^ (英文)Cleland, W.W. The Kinetics of Enzyme-catalyzed Reactions with two or more Substrates or Products 2. Inhibition: Nomenclature and Theory. Biochim. Biophys. Acta. 1963, 67: 173–187.
^ (英文)Price, NC. What is meant by 'competitive inhibition'?. Trends in Biochemical Sciences. 1979, 4 (11): pN272. doi:10.1016/0968-0004(79)90205-6.
^ (英文)Poulin R, Lu L, Ackermann B, Bey P, Pegg AE. Mechanism of the irreversible inactivation of mouse ornithine decarboxylase by alpha-difluoromethylornithine. Characterization of sequences at the inhibitor and coenzyme binding sites. J Biol Chem. 1992 Jan 5;267 (1):150–8. PMID 1730582
^ (英文)Ball, Philip (2006) The Devil's Doctor: Paracelsus and the World of Renaissance Magic and Science. Farrar, Straus and Giroux ISBN 978-0-374-22979-5
^ (英文)Yoshikawa S and Caughey WS. Infrared evidence of cyanide binding to iron and copper sites in bovine heart cytochrome c oxidase. Implications regarding oxygen reduction.. J Biol Chem. May 1990, 265 (14): 7945–7958. PMID 2159465.
^ (英文)Hunter T. Protein kinases and phosphatases: the yin and yang of protein phosphorylation and signaling.. Cell. 1995, 80 (2): 225–236. PMID 7834742.
^ Berg JS, Powell BC, Cheney RE. A millennial myosin census.. Mol Biol Cell. 2001, 12 (4): 780–794. PMID 11294886.
^ (英文)Meighen EA. Molecular biology of bacterial bioluminescence.. Microbiol Rev. 1991, 55 (1): 123–142. PMID 2030669.
^ (英文)Mackie RI, White BA; White. Recent advances in rumen microbial ecology and metabolism: potential impact on nutrient output. J. Dairy Sci. 1 October 1990, 73 (10): 2971–95. PMID 2178174. doi:10.3168/jds.S0022-0302(90)78986-2.
^ (英文)Jennifer McDowall. Enzymes of Glycolysis.. [2015-01-23].
^ (英文)Faergeman N. J, Knudsen J. Role of long-chain fatty acyl-CoA esters in the regulation of metabolism and in cell signalling. Biochem J. April 1997, 323: 1–12. PMID 9173866.
^ (英文)Doble B. W., Woodgett J. R. GSK-3: tricks of the trade for a multi-tasking kinase. J. Cell. Sci. April 2003, 116: 1175–1186. PMID 12615961.
^ (英文)Ciampor F, Cmarko D, Cmarkova J, Zavodska E. Influenza virus M2 protein and haemagglutinin conformation changes during intracellular transport.. Acta Virol. 1995, 39 (3): 171 – 181. PMID 8579000.
^ 預防醫學基金會. 苯酮尿症. [2015-01-23].
^ 台大医院. 紫質症. [2015-01-23].
^ 谢绍安, 祝芷. 着色性干皮症. 眼科新进展. 1984, 3.
^ (英文)Fuhrmann G, Leroux JC; Leroux. In vivo fluorescence imaging of exogenous enzyme activity in the gastrointestinal tract. Proceedings of the National Academy of Sciences. 2011, 108 (22): 9032–9037. PMC 3107327. PMID 21576491. doi:10.1073/pnas.1100285108.
^ Renugopalakrishnan V, Garduño-Juárez R, Narasimhan G, Verma CS, Wei X, Li P. Rational design of thermally stable proteins: relevance to bionanotechnology. Journal of Nanoscience and Nanotechnology. November 2005, 5 (11): 1759–1767. PMID 16433409. doi:10.1166/jnn.2005.441.
^ Hult K, Berglund P. Engineered enzymes for improved organic synthesis. Current Opinion in Biotechnology. August 2003, 14 (4): 395–400. PMID 12943848. doi:10.1016/S0958-1669(03)00095-8.
^ Jiang L, Althoff EA, Clemente FR, Doyle L, Röthlisberger D, Zanghellini A, Gallaher JL, Betker JL, Tanaka F, Barbas CF, Hilvert D, Houk KN, Stoddard BL, Baker D. De novo computational design of retro-aldol enzymes. Science. March 2008, 319 (5868): 1387–91. Bibcode:2008Sci...319.1387J. PMC 3431203. PMID 18323453. doi:10.1126/science.1152692.
^ 88.088.1 Sun Y, Cheng J. Hydrolysis of lignocellulosic materials for ethanol production: a review. Bioresource Technology. May 2002, 83 (1): 1–11. PMID 12058826. doi:10.1016/S0960-8524(01)00212-7.
^ 89.089.1 Kirk O, Borchert TV, Fuglsang CC. Industrial enzyme applications. Current Opinion in Biotechnology. August 2002, 13 (4): 345–351. PMID 12323357. doi:10.1016/S0958-1669(02)00328-2.
^ 90.090.190.2 Briggs DE. Malts and Malting 1st. London: Blackie Academic. 1998. ISBN 978-0412298004.
^ Dulieu C, Moll M, Boudrant J, Poncelet D. Improved performances and control of beer fermentation using encapsulated alpha-acetolactate decarboxylase and modeling. Biotechnology Progress. 2000, 16 (6): 958–65. PMID 11101321. doi:10.1021/bp000128k.
^ Tarté R. Ingredients in Meat Products Properties, Functionality and Applications. New York: Springer. 2008: 177. ISBN 978-0-387-71327-4.
^ Chymosin – GMO Database. GMO Compass. European Union. 10 July 2010 [1 March 2015]. (原始内容存档于26 三月 2015). 请检查|archive-date=中的日期值 (帮助)
^ Molimard P, Spinnler HE. Review: Compounds Involved in the Flavor of Surface Mold-Ripened Cheeses: Origins and Properties. Journal of Dairy Science. February 1996, 79 (2): 169–184. doi:10.3168/jds.S0022-0302(96)76348-8.
^ Guzmán-Maldonado H, Paredes-López O. Amylolytic enzymes and products derived from starch: a review. Critical Reviews in Food Science and Nutrition. September 1995, 35 (5): 373–403. PMID 8573280. doi:10.1080/10408399509527706.
^ 96.096.1 Protease – GMO Database. GMO Compass. European Union. 10 July 2010 [28 February 2015]. (原始内容存档于24 二月 2015). 请检查|archive-date=中的日期值 (帮助)
^ Alkorta I, Garbisu C, Llama MJ, Serra JL. Industrial applications of pectic enzymes: a review. Process Biochemistry. January 1998, 33 (1): 21–28. doi:10.1016/S0032-9592(97)00046-0.
^ Bajpai P. Application of enzymes in the pulp and paper industry. Biotechnology Progress. March 1999, 15 (2): 147–157. PMID 10194388. doi:10.1021/bp990013k.
^ Begley CG, Paragina S, Sporn A. An analysis of contact lens enzyme cleaners. Journal of the American Optometric Association. March 1990, 61 (3): 190–4. PMID 2186082.
^ Farris PL. Economic Growth and Organization of the U.S. Starch Industry. (编) BeMiller JN, Whistler RL. Starch Chemistry and Technology 3rd. London: Academic. 2009. ISBN 9780080926551.
拓展閱讀
總論
詞源與歷史
| 酶結構及作用機理
動力學及抑制
|
| ||||||
| ||||||||||||||||||||||
|